«Псевдо» ли псевдогены

«Я слышал столько клеветы в Ваш адрес, что у меня нет сомнений: Вы — прекрасный человек!»
Оскар Уайльд
Расширение технологических возможностей в биологии заставляет пересмотреть многие устоявшиеся парадигмы. Развитие научных знаний устремляется в сторону многогранности и комплексности взаимодействия элементов биологических систем. Биосистема уже не отдельная, пусть даже и уникальная, снежинка, а настоящий снежок, а возможно, и целый снеговик.
«Раньше было лучше»: трава была зеленее, солнце теплее, колбаса по 2,20 и с «мусорной» ДНК (junk DNA) было все ясно. Размер гаплоидного генома человека 3,2x109 нуклеотидных пар, из него около 1,5 % генома кодирует белки, около 30 % генома кодирует гены, кодирующие белки, остальное — «мусорная» ДНК. А теперь… «Я знаю, что ничего не знаю», — вздыхают генетики-сократики и пересматривают проблему «избыточности» генома.
Псевдогены традиционно рассматриваются как нефункциональные последовательности геномной ДНК, не кодирующие белок. Для обозначения псевдогенов используют символ ψ (исходя из этимологии: от греческого ψευδής — pseudḗs, «ложный, фальшивый, ненастоящий, притворный»), как своеобразное клеймо обманщика. Но заслуженно ли оно?
Несмотря на то, что они широко экспрессируются как в про-, так и в эукариотических организмах, их характеризовали как «геномные окаменелости» или фрагменты «мусорной» ДНК [1].
Псевдогены — эволюционно консервативные элементы, то есть схожие или идентичные последовательности, поддерживающиеся эволюцией, несмотря на процесс видообразования. Такая эволюционная «верность» позволяет предположить их высокую функциональную значимость [2].
По механизмам формирования выделяют процессированные и непроцессированные псевдогены (рис. 1) [3].
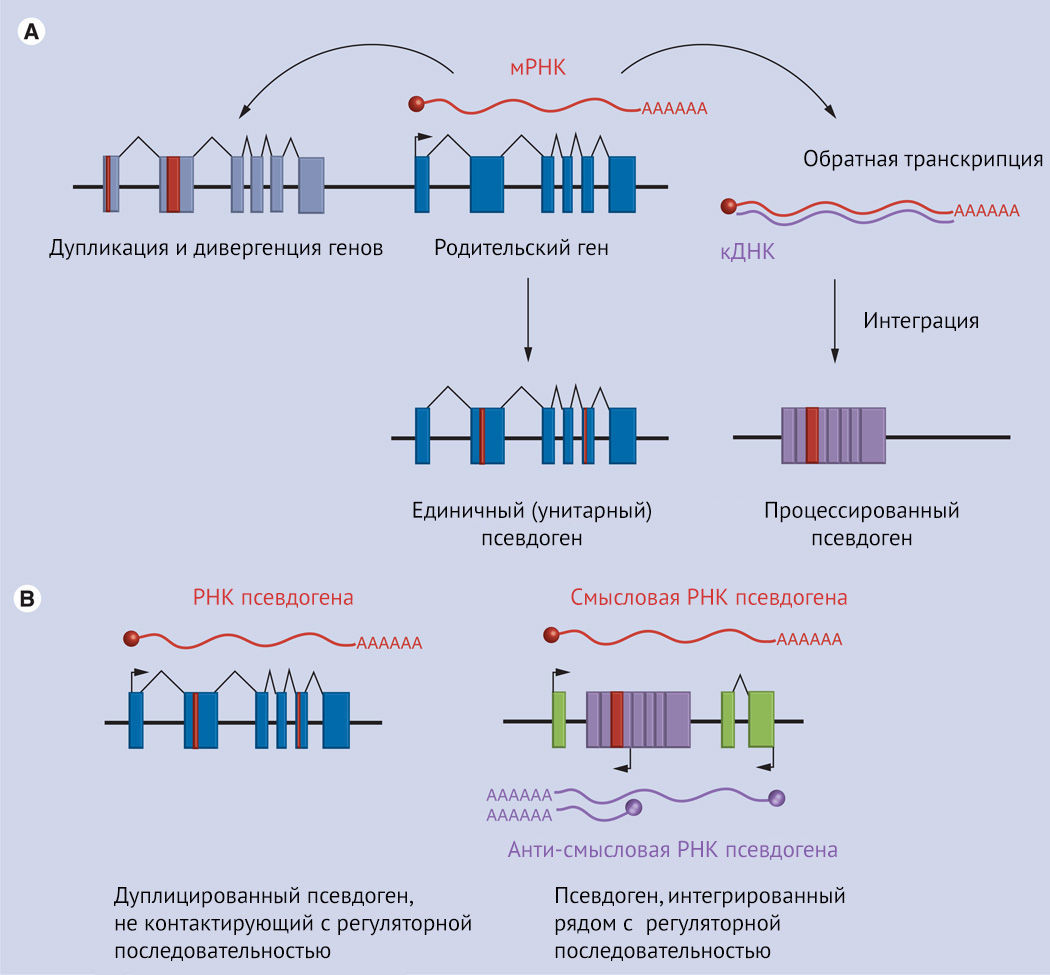
Единичные (унитарные) псевдогены образуются при деградации родительского гена в результате мутаций (обозначено красным).
Дуплицированные псевдогены возникают при копировании локусов гена, часто в сайт, ближайший к родительскому гену. Копия гена впоследствии «псевдогенизируется» путем инактивирующих мутаций.
Процессированные псевдогены образуются при интеграции в геном комплементарной ДНК (кДНК), синтезированной на матрице мРНК при обратной транскрипции. В результате процессированные псевдогены лишены интрон-экзонной структуры.
(В) Транскрибируемые псевдогены (информация с них перезаписывается [транскрибируется] в последовательность РНК). Условием для их возникновения является «захват» контроля над транскрипцией дуплицированными регуляторными последовательностями (т. е. областями промотора или энхансера). Процессированные псевдогены могут интегрироваться в сайты, прилегающие к регуляторным элементам. Транскрипция псевдогена может происходить как в смысловой, так и в антисмысловой ориентации [3].
Процессинг — это комплекс посттранскрипционных модификаций РНК, ее «созревание».
В результате встраивания продуктов обратной транскрипции мРНК соответствующих генов в новый участок генома появляется процессированный псевдоген. В ходе последующих аберрантных мутаций эти копии генов теряют свой кодирующий потенциал. Структурными особенностями таких псевдогенов является отсутствие интронов* и наличие поли(А)-тракта** на 3'-конце.
*Привычная идентификация экзонов как «кодирующих» и интронов как «некодирующих» участков несколько утрирована. Поскольку в ряде случаев интроны (или их части) являются «кодирующими», а экзоны — «некодирующими», точнее называть экзонами те участки ДНК, копии которых составляют зрелую РНК, а интронами — те внутренние районы гена, копии которых удаляются из первичного транскрипта и отсутствуют в зрелой РНК.
**Полиаденилатный хвост образуется в результате посттранскрипционного ферментативного присоединения к 3'-концу мРНК нуклеотидной последовательности, содержащей до 200 адениновых остатков (реакция полиаденилирования).
Также процессированные псевдогены зачастую не содержат промоторов. В таком случае их экспрессия осуществляться с участием других регуляторных элементов [4]. Например, псевдогены, расположенные в интронах других генов, «паразитируют» на аппарате транскрипции гена-хозяина. Приблизительно 10 % генов имеют процессированные псевдогены, чаще всего такой тип псевдогенов имеют гены «домашнего хозяйства» [5].
Эти гены вовлечены в поддержание основ функционирования клетки и, соответственно, экспрессируются на стабильном уровне в разнообразных клетках организма как в нормальном, так и в патологическом состоянии. Такие биологические свойства отвечают критериям идеального контрольного/эталонного гена для изучения экспрессии генов в данном организме [6].
Дуплицированные псевдогены образуются в результате тандемной дупликации или кроссинговера. Последующие мутации делают одну из копий гена функционально неактивной. Такие псевдогены сохраняют интрон-экзонную структуру. В отличие от процессированных псевдогенов, которые могут располагаться в различных участках генома, дуплицированные псевдогены не покидают отчую хромосому и обитают рядом с генами-родителями. Также они не взаимодействуют с сайтами связывания факторов транскрипции, активными маркерами хроматина или сайтами связывания РНК-полимеразы, что подтверждает их функциональную пассивность. Примерами таких инфантильных созданий являются единственный псевдоген в β-глобиновом локусе у людей на хромосоме 11 и три псевдогена в α-глобиновом локусе на хромосоме 16 [7].
В результате повреждающей мутации в единственной копии исходного гена могут возникать одиночные (унитарные) псевдогены. Это псевдогены-«сироты» (тяжелое наследие мутационной нагрузки): они не имеют функционально активного родительского псевдогена [8]. Классическим примером является ген L-глюконо-γ-лактоноксидазы (GULO), продукт которого является ферментом, катализирующим последний этап биосинтеза витамина С (L-аскорбиновая кислота). Этот ген является функциональным у большинства видов позвоночных, потому что витамин С необходим в качестве кофактора в нескольких метаболических реакциях; в частности, обработки коллагена. У приматов он обнаруживается уже в виде неактивного псевдогена [9].
У эукариот внеядерная генетическая информация также может вносить псевдогенные «поправки» в структуру ядерного собрата. Открытие этого феномена могло бы утешить бедного кота Тома, терроризируемого мышонком Джерри, мажора из семьи популярных экспериментальных организмов. Именно домашняя кошка Felis catus стала первым организмом, у которого был описан перенос крупного фрагмента митохондриальной ДНК (мтДНК) размером 7,9 тысяч пар нуклеотидов в ядро. Авторами предложен термин NUMT (NUclear copies of MiTochondrial genes) [10]. Соответственно, NUMT-псевдогены — это фрагменты мтДНК различного размера (как целые митохондриальные гены, так и последовательности двух и более соседних генов), встроенные в различные участки ядерного генома. NUMT-псевдогены — крайне нестабильные структуры и подвержены широкому спектру мутационных изменений: замены, вставки, делеции, дупликации.
Свидетелями защиты псевдогенной значимости выступают следующие факты:
- Многие псевдогены эволюционировали и выжили под давлением естественного отбора, согласующегося с биологической функциональностью.
- Транскрибируемые псевдогены имеют тканеспецифические паттерны экспрессии, в том числе в тканях, в которых нет родительского гена. Например, рецептор 5-HT7 (HRT7) имеет псевдоген, который экспрессируется в печени и почках, не экспрессирующих родительский ген HTR7 [11]. Экспрессия некоторых псевдогенов является динамической величиной и реагирует на физиологические сигналы, такие как клеточный стресс [12].
- Экспрессия псевдогенов обнаружена при различных патологических состояниях, преимущественно при онкологических заболеваниях (табл. 1) [13–20].
Таблица 1 | Экспрессия псевдогенов при различных формах онкологических заболеваний [13–20]
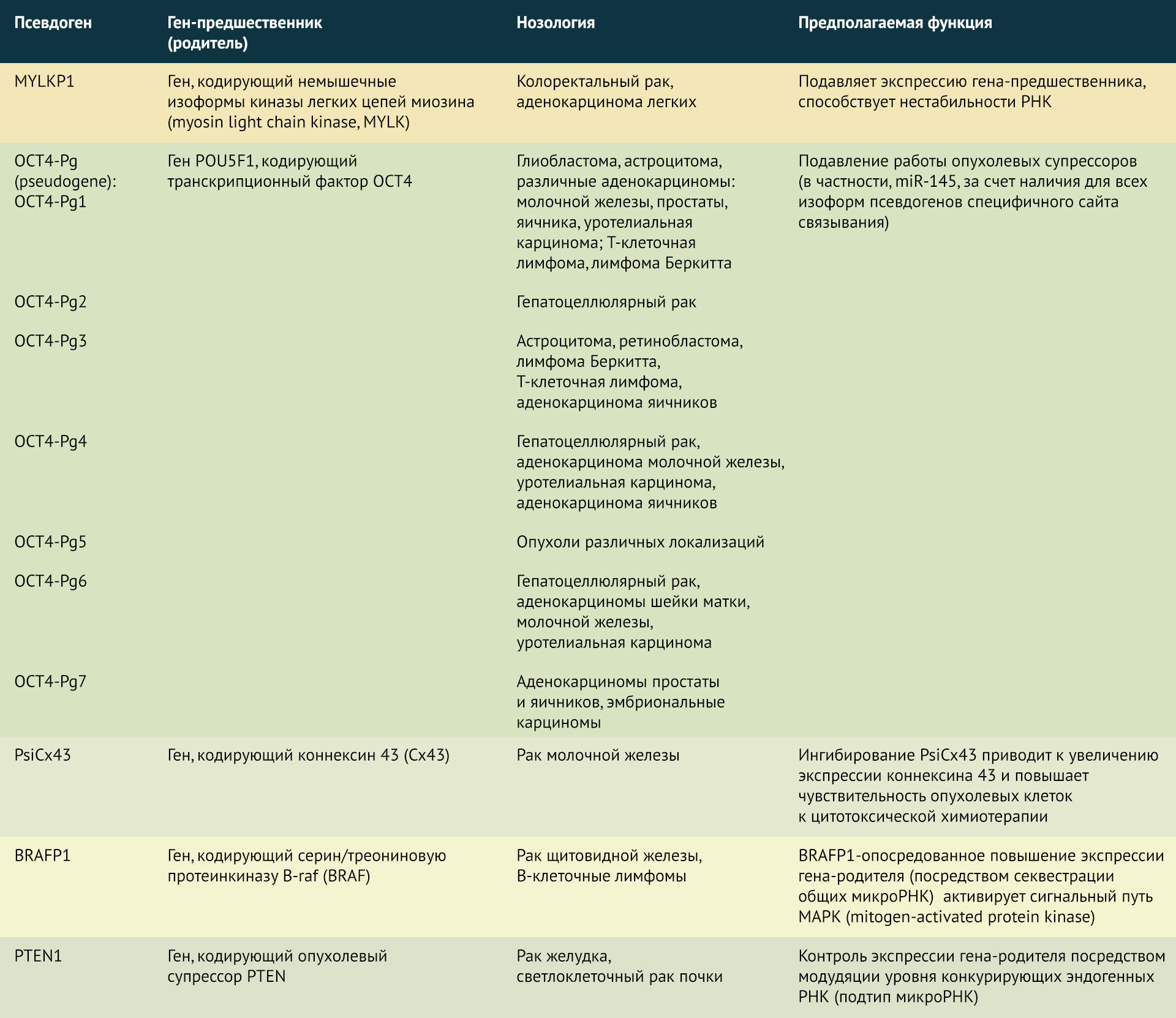
Дифференциальная экспрессия транскриптов псевдогенов отмечена также при патологии иммунного ответа, в частности, при бронхиальной астме [21] и ВИЧ-инфекции [22, 23].
В контексте раскрывающихся возможностей псевдогенов встает вопрос об уместности «псевдо» в их наименовании. Перспективы уточнения роли этих структур для корректного встраивания в генетический пазл связаны с биоинформатическими инструментами. Постоянно нарастающая лавина экспериментальных данных требует систематизации для эффективного использования. Представленная в статье таблица охватывает не столь значительный объем исследований роли псевлогенов в онкологии. Даже силами целого научного коллектива возможно создание весьма ограниченного обзора (относительно всего научного сообщества). Несомненное преимущество для работы с большими данными (BIG DATA) имеют методы in silico (компьютерное моделирование). В результате возможно получить согласованное описание (онтологию) псевдогенов в качестве исчерпывающего источника информации для различных целей.
Помимо включения в общие с «настоящими» генами базы данных (например, https://www.genecards.org/), для работы с псевдогенами разрабатывается, модифицируется и усложняется множество кастомных инструментов. Интересный функционал и качественную поддержку пользователей осуществляют:
- Pseudofam — большая база данных семейств псевдогенов, основанная на базе данных Pfam, которую можно использовать для анализа структуры семейств псевдогенов [24].
- PseudoFuN — комплексная база данных семейств псевдогенов, использующая преимущества вычислений на графическом процессоре [25].
- Pseudo2GO — основанный на графах метод глубокого обучения для прогнозирования функций псевдогена c использованием информации по родственным ему кодирующим генам [26, 27].
Источники:
- Shemesh R., Novik A., Edelheit S., Sorek R. Genomic fossils as a snapshot of the human transcriptome //Proc Natl Acad Sci U S A. — 2006.
- Mahmudi O., Sennblad B., Arvestad L. et al. Gene-pseudogene evolution: a probabilistic approach //BMC Genomics. — 2015.
- Roberts T.C., Morris K.V. Not so pseudo anymore: pseudogenes as therapeutic targets //Pharmacogenomics. — 2013.
- Tang W., Mun S., Joshi A., Han K., Liang P. Mobile elements contribute to the uniqueness of human genome with 15,000 human-specific insertions and 14 Mbp sequence increase //DNA Res. — 2018.
- Reams A.B., Roth J.R. Mechanisms of gene duplication and amplification //Cold Spring Harb Perspect Biol. — 2015.
- Caracausi M., Piovesan A., Antonaros F., Strippoli P., Vitale L., Pelleri M.C. Systematic identification of human housekeeping genes possibly useful as references in gene expression studies //Mol Med Rep. — 2017.
- Yap M.W., Young G.R., Varnaite R., Morand S., Stoye J.P. Duplication and divergence of the retrovirus restriction gene Fv1 in Mus caroli allows protection from multiple retroviruses //PLoS Genet. — 2020.
- Sisu C. et al. Transcriptional activity and strain-specific history of mouse pseudogenes //Nat Commun. — 2020.
- Yang H. Conserved or lost: molecular evolution of the key gene GULO in vertebrate vitamin C biosynthesis //Biochem Genet. — 2013.
- Lopez J.V., Yuhki N., Masuda R., Modi W., O'Brien S.J. Numt, a recent transfer and tandem amplification of mitochondrial DNA to the nuclear genome of the domestic cat //J Mol Evol. — 1994.
- Elliman S.J., Wu I., Kemp D.M. Adult tissue-specific expression of a Dppa3-derived retrogene represents a postnatal transcript of pluripotent cell origin //J Biol Chem. — 2006.
- Kalyana-Sundaram S. et al. Expressed pseudogenes in the transcriptional landscape of human cancers //Cell. — 2012.
- Lynn H. et al. Single nucleotide polymorphisms in the MYLKP1 pseudogene are associated with increased colon cancer risk in African Americans //PLoS One. — 2018.
- Han Y.J., Ma S.F., Yourek G., Park Y.D., Garcia J.G. A transcribed pseudogene of MYLK promotes cell proliferation //FASEB J. — 2011.
- Poursani E.M., Mohammad Soltani B., Mowla S.J. Differential Expression of OCT4 Pseudogenes in Pluripotent and Tumor Cell Lines //Cell J. — 2016.
- Wezel F., Pearson J., Kirkwood L.A., Southgate J. Differential expression of Oct4 variants and pseudogenes in normal urothelium and urothelial cancer //Am J Pathol. — 2013.
- Kalyana-Sundaram S, Kumar-Sinha C, Shankar S, Robinson DR, Wu YM, Cao X, Asangani IA, Kothari V. et al. Expressed pseudogenes in the transcriptional landscape of human cancers //Cell. — 2012.
- Karreth F.A. et al. The BRAF pseudogene functions as a competitive endogenous RNA and induces lymphoma in vivo //Cell. — 2015.
- Zhang R. et al. Long non-coding RNA PTENP1 functions as a ceRNA to modulate PTEN level by decoying miR-106b and miR-93 in gastric cancer //Oncotarget. — 2017.
- Yu G. et al. Pseudogene PTENP1 functions as a competing endogenous RNA to suppress clear-cell renal cell carcinoma progression //Mol Cancer Ther. — 2014.
- Austin P.J.et al. Transcriptional profiling identifies the long noncoding RNA plasmacytoma variant translocation (PVT1) as a novel regulator of the asthmatic phenotype in human airway smooth muscle //J Allergy Clin Immunol. — 2017.
- Gupta A, Brown CT, Zheng YH, Adami C. Differentially-Expressed Pseudogenes in HIV-1 Infection //Viruses. — 2015.
- Devadas K. et al. Analysis of Host Gene Expression Profile in HIV-1 and HIV-2 Infected T-Cells //PLoS One. — 2016.
- Lam H.Y. et al. Pseudofam: the pseudogene families database //Nucleic Acids Res. — 2009.
- Campbell M.J., Huang K., Zhang Y. PseudoFuN: Deriving functional potentials of pseudogenes from integrative relationships with genes and microRNAs across 32 cancers //Gigascience. — 2019.
- Johnson T.S., Li S., Kho J.R., Huang K., Zhang Y. Network analysis of pseudogene-gene relationships: from pseudogene evolution to their functional potentials //Pac Symp Biocomput. — 2018.
- Fan K., Zhang Y. Pseudo2GO: A Graph-Based Deep Learning Method for Pseudogene Function Prediction by Borrowing Information From Coding Genes //Front Genet. — 2020.
