Новые технологии секвенирования

Последнее десятилетие методы секвенирования ДНК активно развивались, становясь быстрее, качественнее и доступнее. Появление новых подходов к секвенированию напрямую влияет на такие области, как персонализированная медицина, генетика [1].
Одномолекулярное секвенирование в реальном времени
В начале 2011 года PacBio выпустила секвенсор PacBio RS. Далее технология продолжила совершенствоваться: средняя длина чтения увеличилась в десять раз, а пропускная способность за цикл — в 100 раз. Уровень «однопроходных» ошибок составляет 13 %, но молекулы до 1–2 кбайт теперь можно многократно секвенировать благодаря циклическим шаблонам и увеличенной процессивности полимераз, что увеличивает точность (рис. 1D) [2]..
В апреле 2013 года компания выпустила улучшенную версию — секвенатор «PacBio RS II», который имеет более высокую пропускную способность и даёт возможность получать более длинные чтения ДНК.
Технология, используемая компанией PacBio — одномолекулярное секвенирование в реальном времени. Эта технология использует замкнутый круговой шаблон однонитевой ДНК, называемый SMRTbell, который создается путем лигирования адаптеров шпильки к обоим концам секвенируемой двунитевой молекулы ДНК (рис. 1A). Праймер и полимеразу присоединяют к адаптору, после чего загружают комплекс в специализированную проточную ячейку, в которой 150 000 пиколитровых лунок, называемых волноводами нулевого режима (ZMW), для PacBio RSII. У более новой платформы Sequel 1 миллион лунок. В каждой ячейке реплицируется одна секвенируемая молекула ДНК [2].
Во время репликации слияние флюоресцирующих нуклеотидов генерирует сигналы, а система камер записывает видео в режиме реального времени, отображая цвет и продолжительность испускаемого света. Диаметр ячейки меньше, чем длина световой волны, что приводит к экспоненциальному затуханию, освещая самое дно ячеек. Это позволяет снизить интерференцию света от соседних дезоксирибонуклеозидтрифосфатов в растворе (рисунок 1B). Время между соединениями нуклеотидов называется межимпульсным интервалом (IPD). Отслеживая его, можно обнаружить различные модификации (рис. 1C). Поскольку SMRTbell образует замкнутый круг, после того, как полимераза реплицирует одну цепь секвенируемой ДНК, она может продолжить использовать адаптер, а затем другую цепь в качестве шаблона. Обе цепи могут быть секвенированы несколько раз в одном непрерывном считывании длинной последовательности, если время жизни полимеразы является достаточным для этого. Затем длинную последовательность можно разделить на несколько более коротких, распознав и вырезав последовательности адаптеров. На рисунке 1D показана связь между количеством проходов и точностью, выраженной в оценке качества (QV). Обратите внимание, что существует связь между длиной молекулы и точностью секвенирования (для более длинных молекул будет меньшее количество проходов) [2].
.
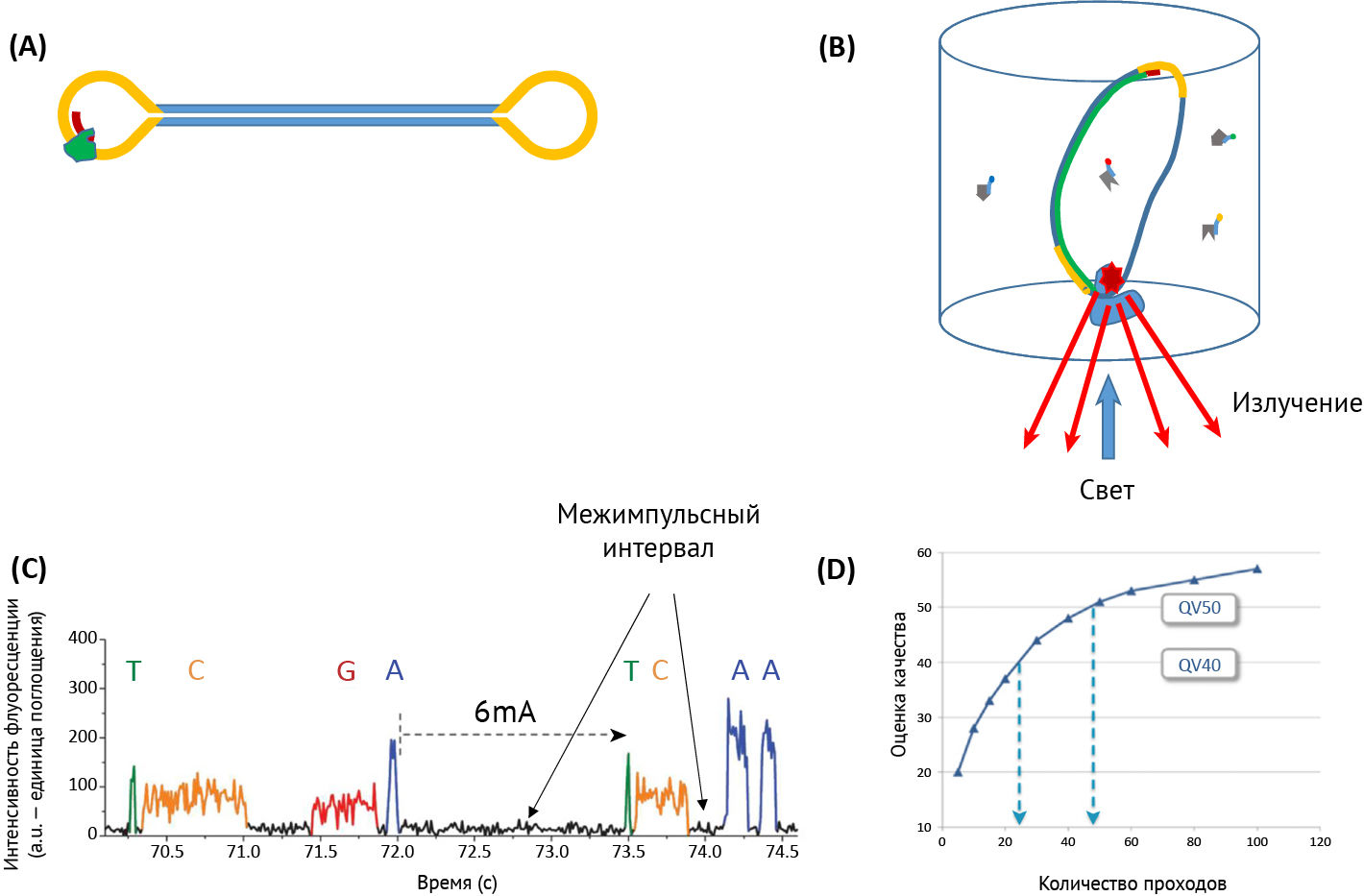
(B) Схематическое представление волновода с нулевой модой (ZMW), наномасштабные камеры наблюдения. Комплекс полимераза-праймер-SMRTbell связывается с дном волновода посредством взаимопритяжения биотина и стрептавидина. Обратите внимание, что не все волноводы будут содержать молекулу ДНК, потому что комплекс загружается путем диффузии. Полимераза включает флюоресцирующие нуклеотиды, испускающие флуоресцентный сигнал при освещении снизу. Эти сигналы записываются камерой в режиме реального времени.
(C) На видео регистрируется не только цвет флуоресценции, но и время между включениями нуклеотидов, называемое межимпульсным интервалом (IPD) (черный). Присутствие эпигенетической модификации, такой как 6-метиладенозин (6-мА), приводит к задержке IPD. Адаптировано с разрешения Pacific Biosciences.
(D) Несколько «проходов» круговой библиотеки можно объединить в кольцевые консенсусные последовательности (CCS), точность которых увеличивается с увеличением количества проходов. Точность выражается как значение качества (QV). Обратите внимание, что при 25 проходах точность достигает 99,999 % (QV40), что аналогично точности секвенирования Illumina. При 50 проходах точность может достигать 99,9999 % (QV50). Адаптировано с разрешения Pacific Biosciences.
Данные, указанные на рисунке, основаны на бактериальном геноме, секвенированном в системе Sequel с использованием программного обеспечения 2.1 и программного обеспечения 5.1.
Секвенирование с использованием нанопор
Идея использования нанопор в мембране для секвенирования одноцепочечной ДНК или РНК возникла в конце 1980-х годов. Но из-за технических сложностей впервые об успешных результатах секвенирования сообщили только в 2012 году [3]. Два года спустя компания Oxford Nanopore Technologies выпустила первый нанопоровый секвенсор [2].
Секвенирование с помощью нанопор происходит в проточной ячейке, в которой два отсека заполнены ионным раствором, разделены мембраной с включенными в нее 2048 (MinION) или 12000 (PromethION) отдельными нанопорами. Изменение постоянного напряжения создает ионный ток через нанопоры, и при транслокации молекулы ДНК или РНК происходят изменения ионного тока (рис. 2A). Ток в нанопоре измеряется датчиком несколько тысяч раз в секунду и графически интерпретируется. Обработка данных выполняется программным обеспечением minKNOW. Секвенирование с использованием нанопор требует подготовки библиотеки, в которой фрагменты ДНК подлежат окончательному соединению с последующим присоединением к адаптеру (рис. 2B). Адаптеры — это комплексы ДНК-белок, тесно связанные ферментом полимеразой или геликазой, они обеспечивают постепенное движение ДНК через поры с помощью специального механизма, обеспечивающего прерывистое вращательное движение нити в одном направлении. Двунитевая ДНК раскручивается в поре, после чего проходит одна нить. Для повышения точности считывания были разработаны механизмы для секвенирования второй нити после того, как первая нить закончила проходить через пору. Для повышения вероятности того, что вторая нить пройдет через пору только после первой, используются специальные последовательности адаптера. Это позволяет считывать информацию с обеих нитей. Первые результаты, демонстрирующие осуществимость секвенирования с использованием нанопор, были получены с использованием пор a-гемолизина (рис. 2C) [2].
Но для точного секвенирования нужны поры с более короткими зонами чувствительности. Первые реальные результаты секвенирования при помощи нанопор были получены с использованием поры MspA. В настоящее время используются поры CsgG [2].
.
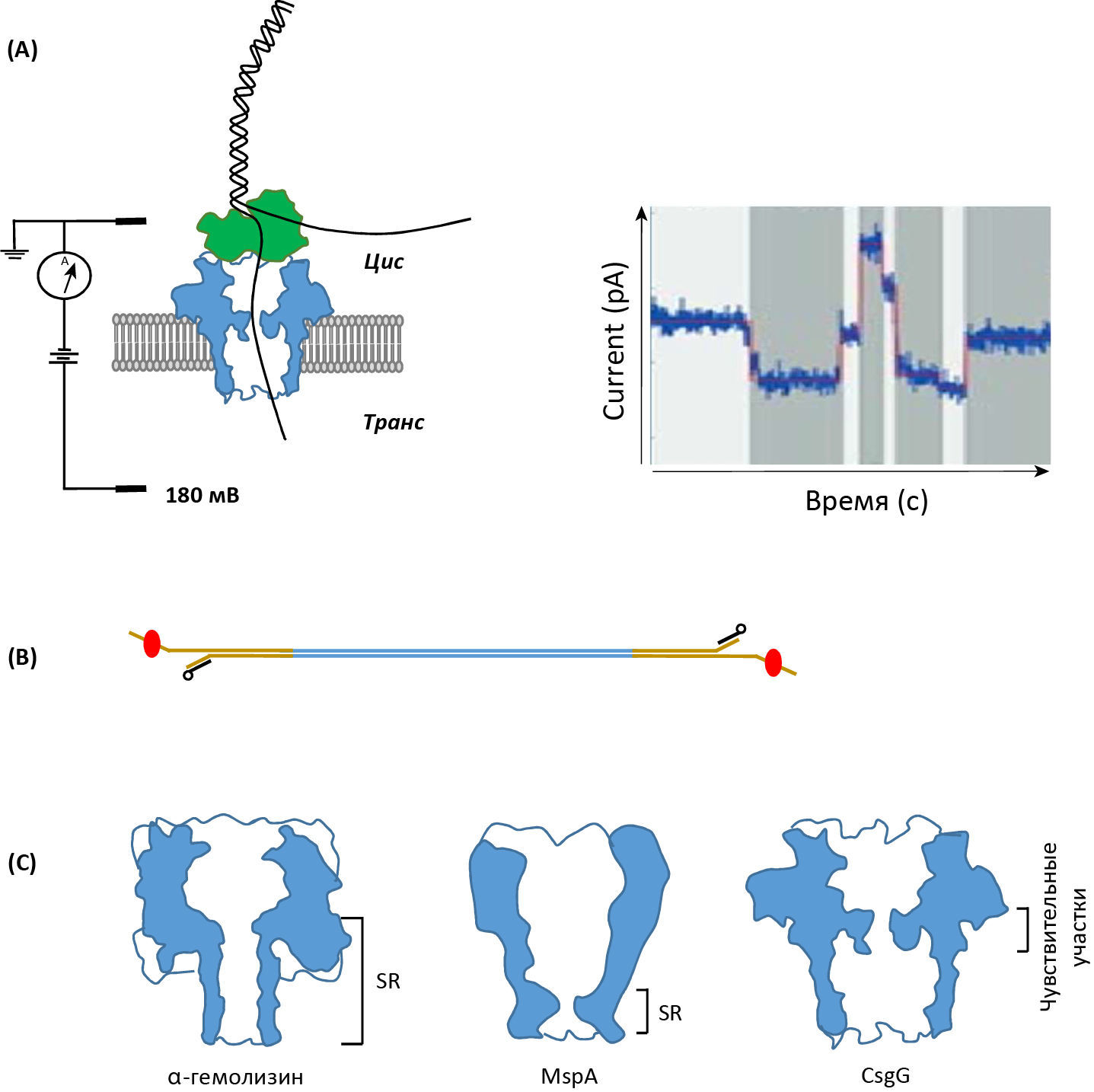
(B) Типичная библиотека. Двухцепочечные фрагменты ДНК (синие) часто проходят необязательный этап репарации ДНК, поскольку одноцепочечные разрывы приводят к преждевременному прекращению секвенирования. Затем происходит подготовка концов для создания подходящих подложек для лигирования адаптеров (коричневого цвета). Адаптеры имеют 50 выступающих концов, с которыми связан «моторный» белок (красный); этот конец первым войдет в пору, и, следовательно, секвенирование происходит в направлении 50-30. С другой цепью адаптера гибридизуется олигонуклеотид с холестериновым фрагментом (черный), который привязывает молекулы библиотеки к мембране и повышает эффективность секвенирования с использованием нанопор.
(С) Различные типы нанопор. Поры а-гемолизина, поры MspA и поры CsgG, которые в настоящее время используются ONT (R9). Указаны короткие чувствительные участки различных пор; обратите внимание, что у пор MspA и CsgG более короткие чувствительный участки, чем у a-гемолизиновых. В результате меньшее количество нуклеотидов вносят вклад в сигнал, что приводит к более точному определению основания.
В отличие от одномолекулярного секвенирования в реальном времени, длина последовательности, считываемой секвенированием с помощью нанопор, ограничена не возможностями метода, а длиной молекул ДНК, которые будут секвенированы. Следовательно, при условии достаточного качества секвенируемой ДНК, могут считываться длинные последовательности (до 1 Мб) [2]. Среди недостатков — высокая вероятность ошибок (15 %), невозможность секвенирования одной и той же цепи несколько раз, как при одномолекулярном секвенирования в реальном времени [4].
Технологии SLR: Illumina / 10X Genomics
Технологии SLR (synthetic long read — синтезируемые длинные последовательности) В отличие от истинных платформ секвенирования с длительным считыванием, основана на системе штрих-кодирования для связывания фрагментов, которые секвенируются на существующих секвенсорах для чтения коротких последовательностей. В настоящее время существуют две системы: платформа секвенирования Illumina SLR (ранее Moleculo; рисунок IA) и система на основе эмульсии 10X Genomics (рисунок Ib) [2].
В системе Illumina геномная ДНК разрезается на фрагменты по 8–10 килобайт, после чего происходит лигирование с образованием «длинного фрагмента». Эти большие фрагменты затем разделяют на микротитрационный планшет (3000 фрагментов в 1 лунке) и подвергают дальнейшей фрагментации и добавлению адаптера посредством тагментации. Каждая лунка содержит один штрих-код. Последовательности ДНК объединяют в пул и проводят секвенирование с последующей локальной сборкой для восстановления исходных длинных фрагментов. С помощью секвенирования на основе эмульсии 10X Genomics природные фрагменты ДНК размером до 100 кб смешиваются в мицеллах в эмульсии вместе с гелевыми шариками, содержащими последовательности адаптера и штрих-кода (рисунок Ib). Благодаря микроструйной технологии 10X Genomics требуется очень небольшое количество исходного материала (1 нг). В каждой мицелле шарик геля растворяется, и более мелкие фрагменты ДНК амплифицируются из исходных больших фрагментов, каждый со штрих-кодом, идентифицирующим исходную мицеллу. Затем фрагменты со штрих-кодом объединяют, а затем проводят классическую подготовку и секвенирование библиотеки Illumina. Полученные последовательности собирают, чтобы сформировать серию стабильных фрагментов по 50 кб [2].
.
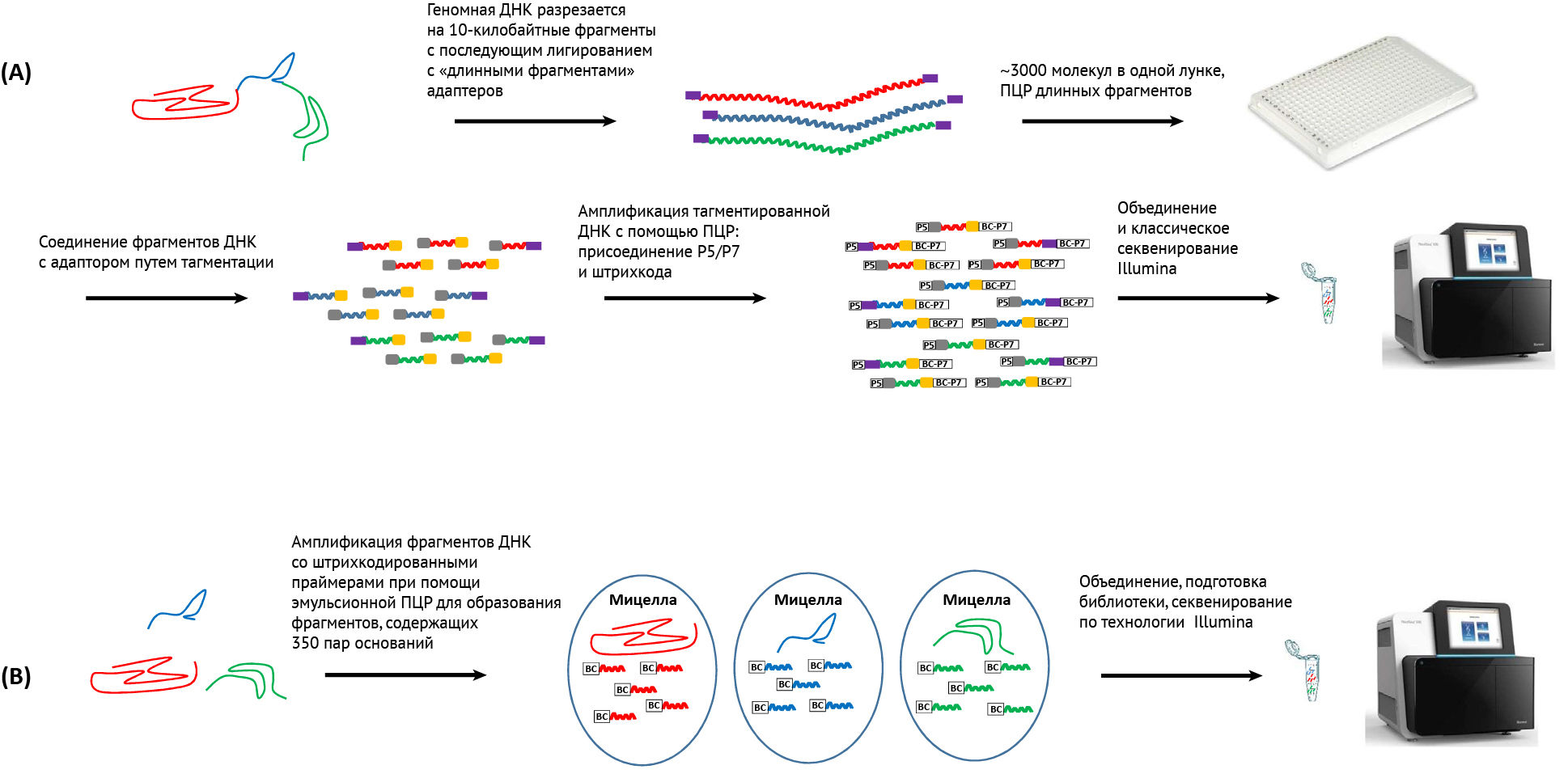
(B) 10X Genomics — эмульсионное секвенирование. Прибор Chromium может разделять природные фрагменты ДНК на 1 миллион мицелл («GEM») вместе со штрих-кодированными праймерами (доступно 4 миллиона штрих-кодов) и реагентами для ПЦР. В каждой мицелле меньшие фрагменты ДНК амплифицируются из исходного большего фрагмента, каждый со штрих-кодом, идентифицирующим исходную мицеллу. Получающиеся меньшие фрагменты объединяются и секвенируются по механизму Illumina. После секвенирования последовательности упорядочены и связаны друг с другом, образуя серию стабильных фрагментов в диапазоне 50 КБ.
Быстрота и дешевизна методов секвенирования нового поколения, недоступная ранее, спровоцировала бум в индустрии геномных исследований. Появилась возможность проводить ранее технически недоступные эксперименты.
Источники:
1. Levy S. E., Boone B. E. Next-Generation Sequencing Strategies //Cold Spring Harbor perspectives in medicine. – 2019. – Т. 9. – №. 7. – С. a025791.
2. van Dijk E. L. et al. The third revolution in sequencing technology //Trends in Genetics. – 2018. – Т. 34. – №. 9. – С. 666-681.
3. Manrao E. A. et al. Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase //Nature biotechnology. – 2012. – Т. 30. – №. 4. – С. 349.
4. Jain M. et al. MinION analysis and reference consortium: phase 2 data release and analysis of R9. 0 chemistry //F1000Research. – 2017. – Т. 6.
